5.3 分析生物衰老学中的基因表达
生物学上最重要的进步之一发生在20世纪70年代,当时科学家能够从染色体上去除一段DNA并确定其序列。提取和操控DNA片段的能力使得技术得到发展,让科学家能够操纵基因序列或开发全新的基因,然后研究这些处理如何影响细胞和生物体的功能。这些技术,统称为DNA重组技术,将改变所有生物学研究的方向。从生态学到分子遗传学,科学家们现在可以识别特定的基因,并且更全面地了解生命的基本机制。
重组DNA技术也催生了商业生物技术产业的繁荣。曾经只有最训练有素、技术最娴熟的科学家才能使用的技术现在已经实现了自动化和商业化,因此非专业人士可以将复杂的基因分析技术应用于特定的研究兴趣。生物学家开始使用这些技术来识别可能影响衰老速率和寿命的基因。
在第1章中,您了解了生物衰老学家如何利用酵母、蠕虫和果蝇等简单生物来研究衰老的遗传学。这些生物为生物衰老学家提供了几个优势,包括(1)它们的基因组已经测序,即所有基因的序列和位置都是已知的;(2)利用这些生物进行一般遗传学研究的悠久历史,已经产生了数百甚至数千种具有多种基因型和表型的突变体;(3)这些生物的寿命很短,允许在一个月内(酵母)或一年内(蠕虫和果蝇)进行几次寿命研究。你还了解到,在这些简单的真核生物中,任何被鉴定为调节衰老和寿命的基因都会有基因同源基因,根据系统发育关系的理论,在更复杂的生物中具有相似的功能。一旦一个基因在简单的真核生物中被证明影响寿命,就可以在更复杂的生物(如小鼠)中鉴定出类似的基因,并评估其与人类衰老或寿命的可能相关性。
利用对这三种模式生物的遗传学积累的大量知识,生物衰老学家发现了一些似乎直接调节衰老速率和寿命的基因。这些发现的细节将在下面的章节中介绍。首先,我们探索了发现这些长寿基因的一般技术和方法。
识别调节衰老速率和寿命的基因的一般过程需要几个步骤(图5.15)。这个过程的复杂性使得没有一个研究人员或实验室能够完成所有所需的步骤。基因发现过程反映了全世界的研究人员群体,他们依靠彼此的科学能力来执行过程中的关键步骤。尽管这里简要描述的技术和方法是作为一个单一的、无缝的过程来介绍的,但请记住,每一个步骤在很大程度上都需要独立的实验室在几年内完成工作。
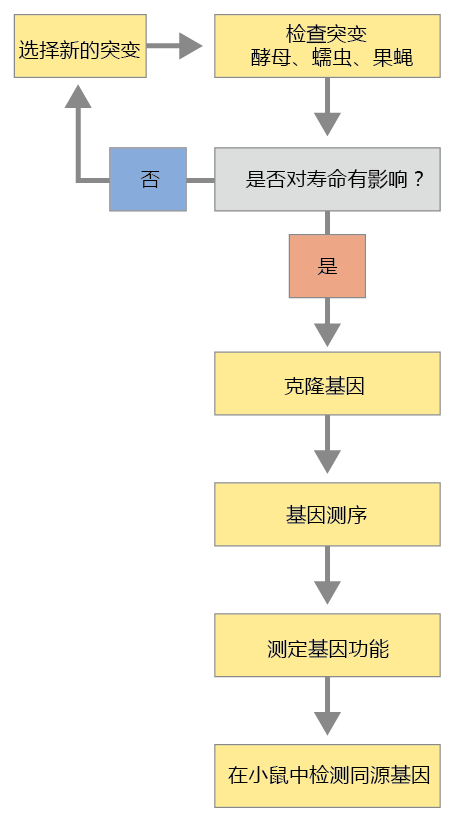
图5.15 识别影响寿命的基因的一般方案。研究人员查阅计算机数据库或研究文献,选择具有可能影响寿命的基因型或表型的突变株。一旦选择了突变株,下一步就是克隆基因。克隆是制作几个相同拷贝的过程,以便有足够的遗传物质对基因进行测序。一旦基因被测序,其功能就被确定了。该过程的最后一步是在小鼠中鉴定该基因的同源性,并评估其是否对长寿有类似的影响。
生物衰老学中的遗传分析始于突变体的筛选
到目前为止,在衰老研究中使用酿酒酵母、秀丽酵母和果蝇进行基因分析的最大优势是,它们的整个基因组都已测序。然而,基因测序并不能提供有关该基因功能的信息。为了获得这一知识,遗传学家首先创建这些生物的突变株,其中单个基因被改变以产生特定的基因型。观察突变株和野生型(即处于正常或自然状态的生物体,不同于非典型或突变型)基因型之间的差异,科学家们第一次了解了该基因的功能。生物衰老学家先假设某个基因如何影响衰老速率和长寿,然后选择这个基因的突变株,最后看看基因型是否与假设一致。(因为大多数研究都是用公共资金进行的,所以研究人员有义务将他们的突变株提供给其他科学家。)一般来说,生物衰老学家选择具有影响生长、繁殖或两者的突变株。(回顾第3章,生长、繁殖和寿命之间的密切关系。)由于许多基因影响生长和繁殖,生物衰老学家必须选择几种不同的突变体,然后比较它们的寿命特征,以确定哪种最适合他们的需要。
突变菌株的比较,即基因筛选,似乎相对容易和直接。然而,这些基因分析的开始步骤是最关键和最困难的。大多数基因都是多效性的(单个基因产生一种以上表型),单个基因突变了,但是突变株通常表现出好几种表现型的改变。即使其中一个突变菌株具有理想的延长寿命的作用,但是它也可能表现出相反的表现型,从而混淆了结果。因此,遗传生物衰老学家可能需要对突变进行细化,以便只存在长寿表型。要细化基因组,使一个基因突变只产生一个基因型或表型,需要进行细致的研究,并进行大量的尝试和错误,最终以失败告终。研究人员可能需要数年时间才能成功开发出一种突变体,为测试基因对长寿影响的假说提供了一个合适而可靠的实验系统。
基因功能的鉴定需要DNA克隆
即使在科学家开发出具有延长寿命的表型的可靠突变株后,该基因的功能和调控以及其编码的蛋白质的结构仍然是未知的。为了确定基因的功能,必须克隆它,也就是说,必须创建基因的相同拷贝。通过克隆基因,研究人员可以产生足够的DNA材料来确定基因的碱基对序列。了解碱基对序列,研究人员可以确定其编码的蛋白质的氨基酸序列,从而确定该蛋白质的可能功能。
克隆过程曾经是一项艰巨的任务,需要几个不同的程序,可能需要几天才能完成。如今,这些程序是自动化的,并结合成一个称为聚合酶链式反应(PCR)的单一过程,只需几个小时即可完成(图5.16)。这个过程开始于从组织或细胞中分离DNA或mRNA,过程相当简单。如果科学家需要克隆基因的整个基因组序列,则只能测DNA而不能从mRNA开始,因为基因组DNA包括外显子(mRNA编码序列)和内含子。如果确定蛋白质的氨基酸序列是目标,则只需要基因的外显子序列,起始材料是mRNA(记住mRNA是仅外显子的互补产物)。这里我们描述了使用mRNA作为起始材料的过程。
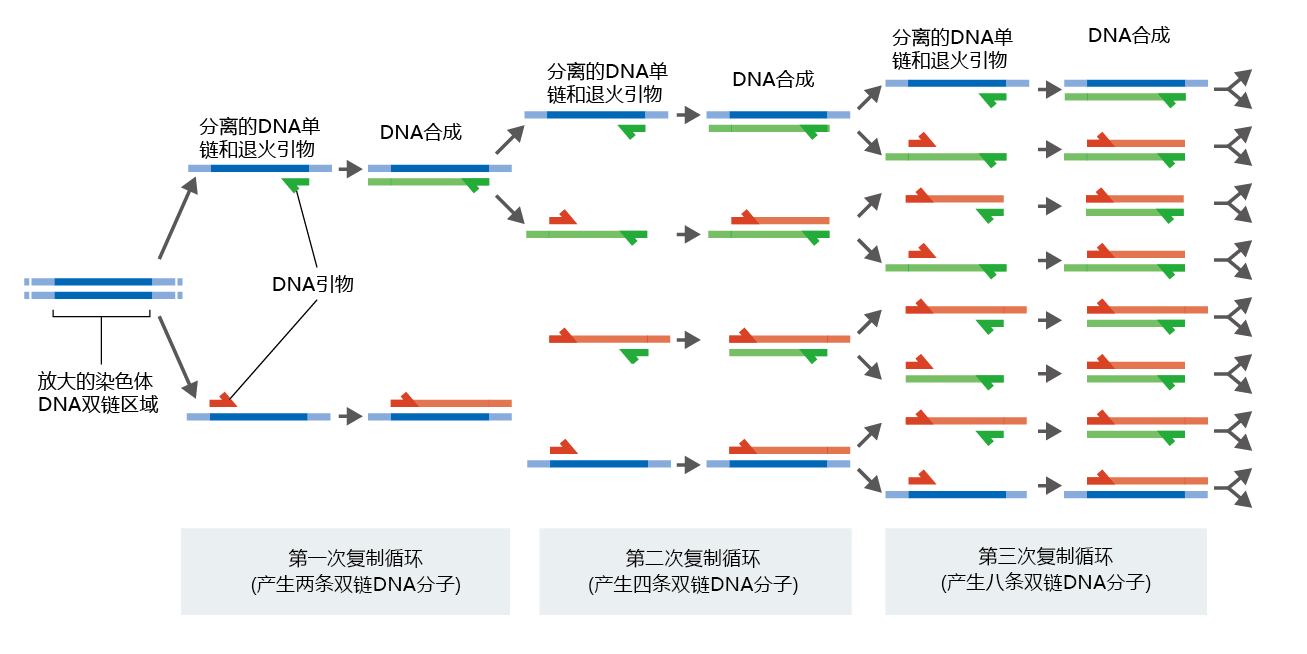
图5.16 使用PCR克隆基因。在鉴定出要克隆的DNA片段后,将DNA加热以分离链,并将短的互补序列(称为引物)连接到待复制序列的3′端。引物将逆转录酶引向基因的起始点(译者注:原著用到了逆转录酶,但是如果模板链没有用到mRNA的话基本不需要逆转录酶)。然后将DNA与DNA聚合酶和脱氧核糖核苷酸三磷酸混合,复制DNA以产生两个子DNA分子。多次重复这个过程,使用新合成的片段作为模板,可以快速放大拷贝数。
在开始PCR过程之前,研究人员设计并用化学方法制造一组寡聚核苷酸(寡聚核苷酸,意为“许多”),这些寡聚核苷酸与分离的基因的3′端完全互补。基因的3′端是已知的,因为用于开发突变体的基因序列是已知的。在日常实践中,研究人员设计寡聚核苷酸,并将这些计划发送给一家公司进行合成。当寡聚核苷酸或“引物”被添加到含有mRNA的溶液中时,它们会引导一种名为逆转录酶的酶(从mRNA中提取DNA,与细胞中通常的过程相反)开始复制被扩增的基因。制造DNA所必需的脱氧核糖核苷酸也被添加到溶液中。
逆转录酶使用mRNA模板制作一条新的互补DNA(cDNA)链,该链只包含被分离基因的外显子。
在这个过程中,mRNA和新的cDNA通过双链氢键结合在一起,必须分离,以便只扩增cDNA。PCR机器将溶液加热至约90°C以打破氢键。然后添加DNA聚合酶和与cDNA 3′端互补的第二组引物,并制备与第一条cDNA互补的新的cDNA链。PCR机器缓慢冷却溶液,两条互补的cDNA链形成双螺旋。扩增过程现在只需在脱氧核糖核苷酸和DNA聚合酶存在下加热和冷却溶液即可。每个周期都会使相同基因的数量加倍。大约需要30个周期才能产生足够的基因测序材料。
基因的功能可以从其序列中部分确定
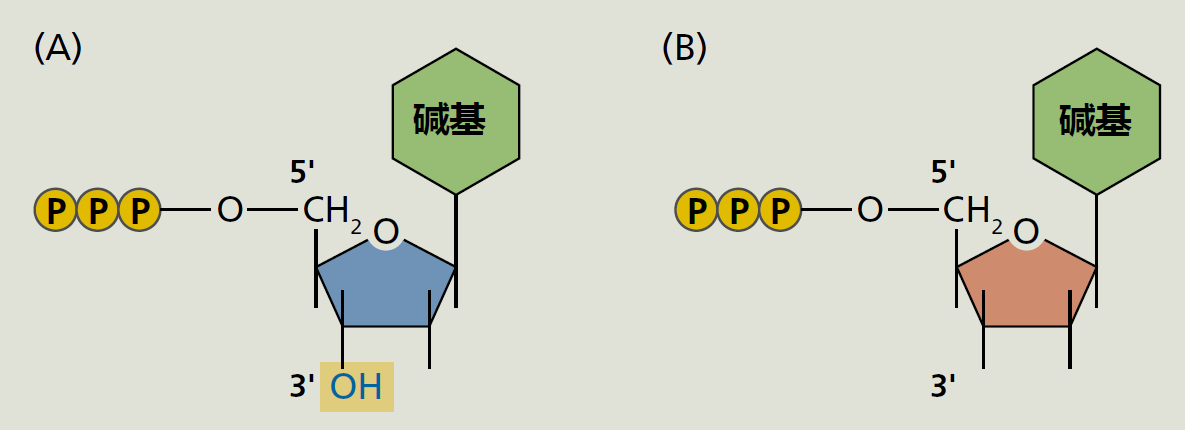
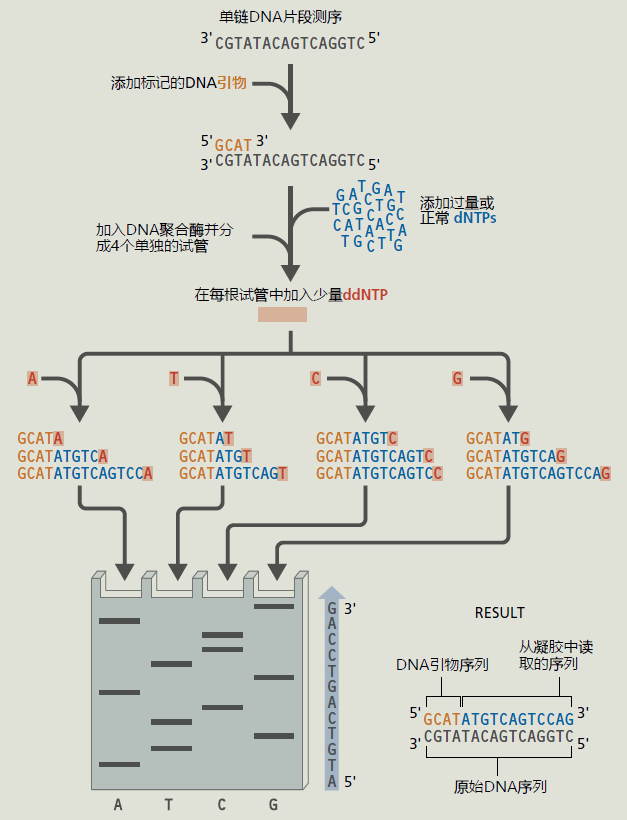
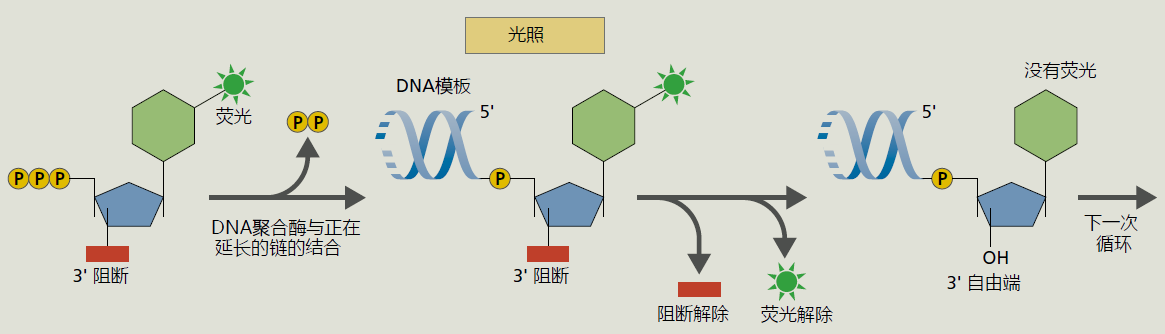
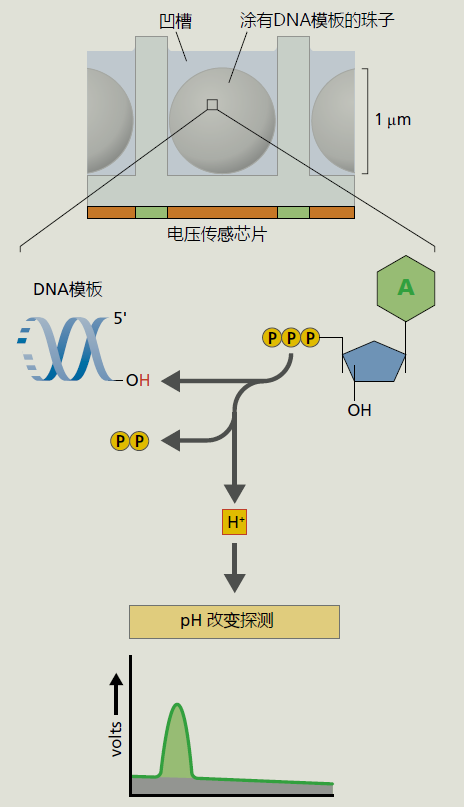
与PCR一样,基因测序现在是自动化的(方框5.2)。将新合成的基因拷贝添加到一种溶液中,该溶液将cDNA分解成终止于特定碱基的片段。因为新基因有数十亿个拷贝,所以cDNA片段会在基因的每个位置终止。测序机器的机器人混合试剂,将荧光染料添加到cDNA片段的末端碱基。每个碱基都标有不同的颜色。使用凝胶电泳(图5.24),通过原子量分离片段,该电泳也由测序机自动进行。基于荧光染料的存在,测序仪中的检测器读取凝胶上每个片段相对于其他片段的位置,并在计算机的帮助下,测序器确定基因的序列。
| 框5.2 基因组测序 |
|
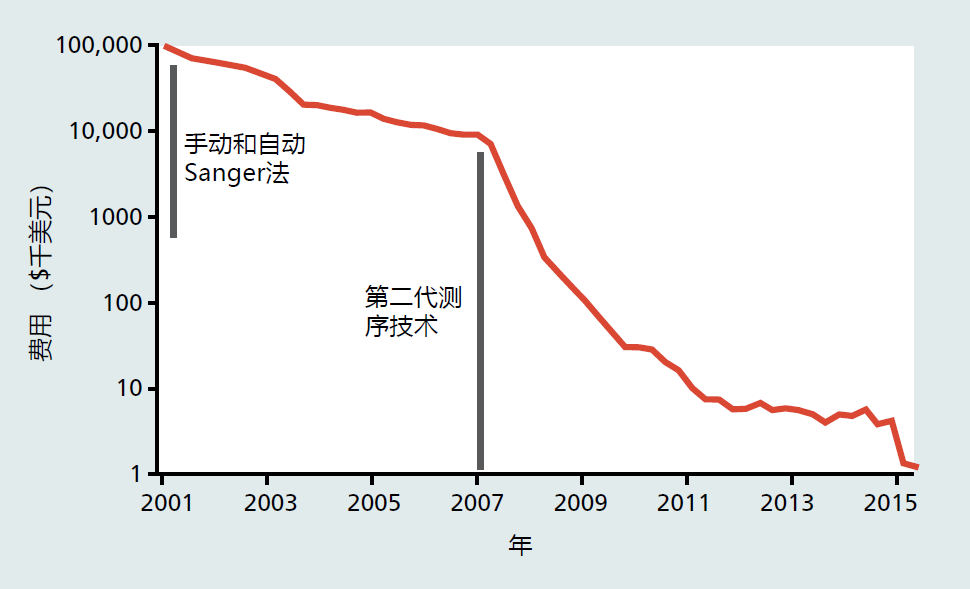
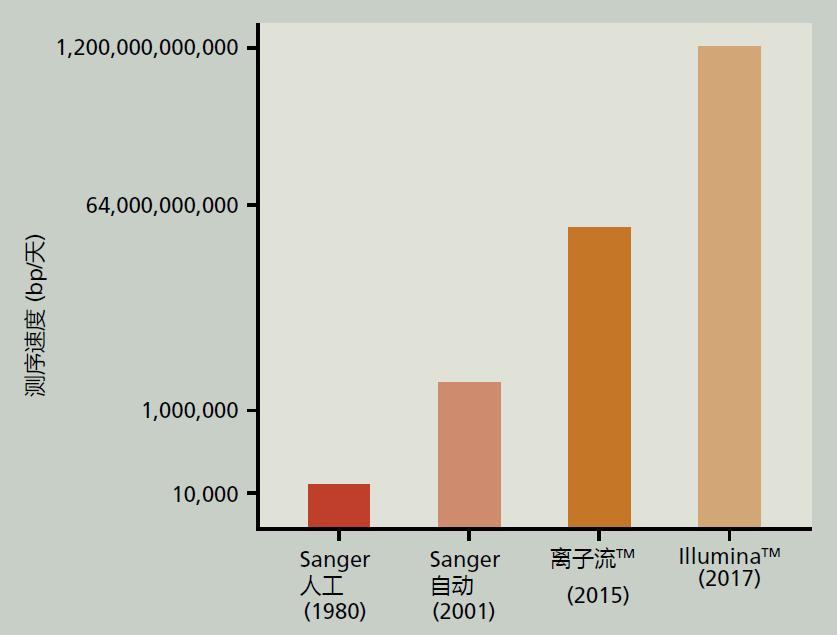
您在第2章中了解到,实现精准医学计划的目标在很大程度上取决于快速、廉价地对每个人的独特基因组进行测序的能力(PMI;框2.2)。美国国家人类基因组研究所(美国国家卫生研究院的一部分)建议,基因组测序必须低于每人1000美元,才能成为临床常规。我们正在迅速实现这一目标,因为人类基因组测序的成本目前仅为1000美元多一点,大约需要24小时(图5.17);当本版书出版时,人类基因组测序的成本很可能低于目标价格。这是一项相当了不起的成就,因为人类基因组的第一个序列在经过13年的重点研究后于2003年完成,耗资5-10亿美元。
图5.17 2001年至2016年人类基因组测序成本(Wetterstrand KA.DNA测序成本:来自NHGRI基因组测序计划(GSP)的数据)。 在这里,我们简要回顾了DNA测序技术的过去、现在和未来。由于DNA测序的所有方面都无法在这篇简短的综述中涵盖,因此需要一些知识上的飞跃。例如,目前常用的测序方法要求将DNA的完整链分割成数万个不同的片段。人类基因组的30亿个碱基对(bp)对于目前的方法来说太大了,无法将它们首尾相连地排列成一个片段。常见的方法还需要扩增原始DNA片段,即克隆,以便为测序提供足够的材料。人类已经建立完善的片段化和扩增方法,这里没有涉及(参见“基因功能的鉴定需要DNA克隆”一节,了解每种方法的简要描述)。此外,将片段映射到其在染色体上的精确位置的技术也没有讨论。
|
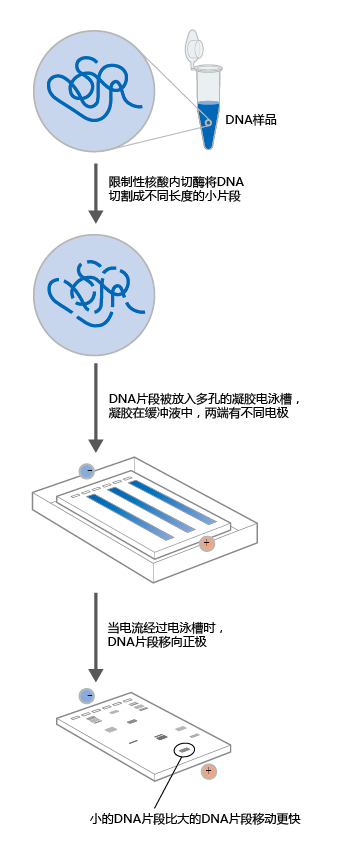
图5.24 凝胶电泳。多孔凝胶上的电泳根据DNA分子的大小和电荷量物理性的分离DNA分子。
该过程的最后一步涉及将遗传密码(见表5.1)应用于基因序列,以确定其编码的蛋白质的氨基酸序列,从而确定其可能的功能。然而,原始基因序列并不能告诉我们编码特定氨基酸的第一组三个碱基(即起始密码子)从哪里开始。在这一阶段,研究人员只能根据以前的遗传学知识,对基因的功能进行预测。美国国立卫生研究院拥有一个大型计算机数据库,其中包含已知功能的基因序列。(一旦确定了基因的功能,研究人员就将其基因序列提交到该数据库中。)计算机软件已经开发出来,可以让研究人员将其序列与已知基因序列进行比较,并推断他们发现的基因可能功能的信息。研究人员可以利用这些信息来集中研究,利用重组技术,可以更精确地确定基因的某些特征,包括起始密码子和终止密码子。
原位杂交可以揭示基因的功能
确定基因的功能通常从评估基因表达的位置和时间开始。为了实现这一点,可以使用一种叫做原位杂交的技术(in situ是拉丁语中“就地”的意思)。由于基因的序列是已知的,因此可以用可检测和可测量的化合物(例如放射性同位素或荧光染料)标记基因片段,并将其引入细胞、组织或整个生物体。当这些标记的核酸探针遇到mRNA的互补序列时,它们与该序列结合并产生杂交分子。然后可以使用适当的检测机器分离和测量杂交分子。
原位杂交是另一种可用于评估基因表达模式的技术。例如,假设我们使用计算机数据库确定了从秀丽隐杆线虫中提取的基因最可能的功能。我们的分析表明,该基因的序列与在特定环境温度下表达mRNA的基因具有高度同源性。因为我们的基因是独一无二的,我们不知道温度可能是多少。我们可以通过向秀丽隐杆线虫注射标记的DNA探针,然后将动物暴露在不同的温度下,用这种方法来确定温度,从而确定基因的功能。当达到导致最大基因表达的温度时,将发生最大的原位杂交。此外,该实验还能确定哪些细胞或组织表达该基因的量最多。如本章稍后所述,后面还有一个实验,用来确定秀丽隐杆线虫中的哪些细胞表达与延长寿命相关的蛋白质。
遗传改变的生物有助于评估基因对人类寿命的影响
回想一下,遗传衰老学家开始对简单生物进行基因分析的众多原因之一是,它们比更复杂的生物寿命更短。这意味着科学家可以在相对较短的时间内筛选长寿基因。一旦一个基因被克隆和测序,并在简单的生物体中确定其功能,就可以开发出生理学上更接近人类的更复杂动物的突变株。总的来说,老鼠一直是进行衰老和寿命遗传分析的首选哺乳动物。
评估基因对衰老或寿命的影响的一种常见方法是开发具有额外基因拷贝或不具有该基因的突变型小鼠。将这些突变体与野生型小鼠进行比较,可以证明该基因是否会改变衰老或寿命。在确定了要修改的小鼠基因后,有必要知道基因表达或基因沉默是否影响寿命的增加(从对简单生物的分析中获得的知识)。如果抑制(基因沉默)表达可使寿命加长,那么基因同源物就会从小鼠基因组中删除。这种类型的突变体被称为基因剔除(knockout)。如果该基因的表达使寿命延长,那么添加额外的基因会使小鼠比野生型小鼠更高的表达。这种类型的突变体被称为转基因生物。
移除或插入一个基因以创造一种突变的小鼠,虽然在当今的研究环境中很常见,但这曾经是一个费力的过程,需要通过几个月的杂交后代得到。2012年,随着一种基因编辑方法的引入,这一切都发生了变化,该方法使用了一种在细菌中发现的防御机制,称为CRISPR(有序排列的回文重复序列[发音为“crisper”])(图5.25)。小鼠的突变株现在可以在一代繁殖中培育出来,即3周。
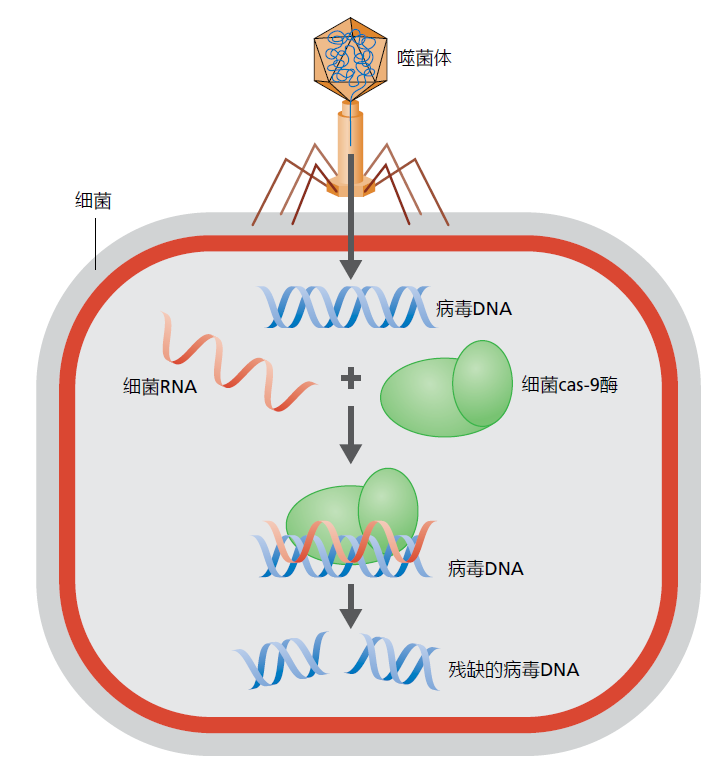
图5.25 细菌对病毒感染的防御系统CRISPR。当噬菌体(一种感染细菌的病毒)将其DNA注入细菌时,细菌识别该序列并产生与病毒DNA序列互补的RNA。RNA与核酸酶Cas-9结合,一起寻找并连接到病毒DNA中的匹配序列。一旦连接,Cas-9切割病毒DNA,使其失效,从而防止病毒感染。
当科学家观察到某些细菌对病毒感染免疫时,CRISPR被发现。令人惊讶的是,这种免疫力具有进化基础。也就是说,细菌在自己的DNA中储存了一份记录,记录了感染前几代的病毒DNA序列。这种“DNA历史”使细菌能够检测到病毒DNA的存在并启动免疫反应。当病毒将其DNA注入细菌时,细菌会产生与病毒DNA互补的RNA分子(图5.25)。互补RNA与核酸酶(一种切割DNA的酶)结合,开始“搜索”病毒DNA。细菌RNA序列被称为向导RNA(guide RNA)。一旦被发现,向导RNA就附着在病毒DNA上,而细菌核酸酶Cas-9通过切割病毒的DNA使病毒失效。
科学家们很快认识到CRISPR–Cas-9系统可以用作DNA编辑工具,以快速开发小鼠的突变株。例如,也许我们想开发一种转基因小鼠,该小鼠过度表达一种延长秀丽隐杆线虫寿命的基因(图5.26)。使用实验室中的分子生物学工具,设计了与小鼠基因同源物的DNA序列互补的向导RNA,并将其连接到Cas-9酶上。(Cas-9核酸酶是从细菌菌落中分离出来的。)RNA同源序列还包含两到六个碱基,与紧邻基因起始端的DNA序列相匹配,该序列称为原间隔区相邻基序(PAM)。然后将含有RNA和Cas-9的CRISPR复合物与克隆基因的DNA序列一起注射到小鼠胚胎中。CRISPR–Cas-9分子找到匹配的DNA序列,解开螺旋,并在PAM处连接到DNA的单链。Cas-9核酸酶切割DNA,克隆的基因附着上去。新的单链序列被用作同源重组的模板,插入的基因成为生物体基因组的一部分。我们现在有了一个胚胎,它有一个额外的基因拷贝,据报道可以延长寿命。然后使用体外受精方法使小鼠受孕,在小鼠怀孕期的3周内,我们成功地培育出了转基因小鼠。
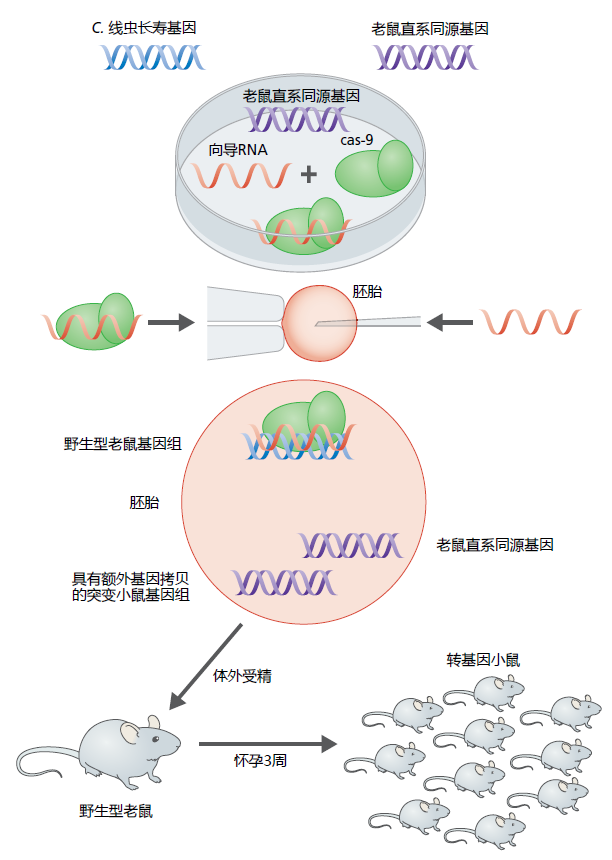
图5.26 用于开发衰老研究转基因小鼠的CRISPR–Cas-9方法的简要说明。计算机搜索确定了秀丽隐杆线虫长寿基因的小鼠基因同源性。然后,使用实验室方法设计小鼠基因的向导RNA,并将其连接到Cas-9核酸酶上。将向导RNA/Cas-9复合物和小鼠同源DNA的单链拷贝注射到小鼠胚胎中。向导RNA定位小鼠基因中的特定序列,并在DNA中进行切割,从而插入小鼠同源基因。使用单链DNA作为模板,同源重组修复了切割,该基因的额外拷贝现在是小鼠基因组的一部分。也可以以类似的方式创建基因剔除小鼠,方法是只向胚胎注射向导RNA–Cas-9复合物,并允许非同源重组在基因移除后修复切割。
DNA微阵列用于评估不同年龄的基因表达模式
我们此处的讨论集中在用于确定单个基因功能的重组DNA技术上。然而,在现实世界中,一个细胞可能会对单个刺激或细胞事件做出反应,表达数百甚至数千个基因。直到20世纪90年代中期,确定对单一刺激的基因表达模式是不可能的。杂交技术的出现使得一种名为DNA微阵列的工具被开发出来,该工具允许研究人员在单个时间点确定细胞的基因表达模式。
DNA微阵列是一块玻璃,大约有邮票大小,嵌入了数千个单链DNA序列,这些序列来自具有已知或未知功能的特定基因。机器人被用来将DNA序列精确地放置在玻璃上,以便知道它们的位置。然后,从待测细胞中提取mRNA并逆转录出cDNA,这些cDNA用荧光探针标记。微阵列和cDNA探针一起孵育,以便让杂交发生。然后清洗微阵列以去除未结合的探针,并使用自动显微镜通过荧光识别杂交分子。图5.27显示了这一过程,使用的是两个不同年龄小鼠肌肉细胞中分离的mRNA。

图5.27 DNA微阵列显示了基因表达模式。DNA微阵列通常用于评估用不同刺激处理或在不同条件下获得的两个样品中的基因表达模式。在这个假设的例子中,mRNA是从两个不同年龄的小鼠的肌肉细胞中分离出来的。将mRNA逆转录成cDNA后,用红色(年轻小鼠)或绿色(老年小鼠)荧光染料标记片段并混合在一起。溶液可以让微阵列上的cDNA片段杂交。洗涤掉未结合的片段后,用自动检测器扫描荧光。绿色圆点表示老年小鼠的基因表达更大;红点圆点表示在年轻小鼠中表达更多;黄点表示在年轻和年老小鼠中均有表达;褐色点表示两组均未表达该基因。(由Alila Medical Images/Shutterstock提供)
在已经完成的类似于图5.27所示假设示例的实验中显示,结果好坏参半。年轻(3–6个月)和年老(26–30个月)小鼠之间基因表达的差异非常小,并且取决于从中分离mRNA的器官。对于所有器官,年轻和年老小鼠之间的差异都没有超过微阵列上显示的总基因的3%,大多数器官显示的差异仅为1%-1.5%。这些值在很大程度上被认为是统计上不重要的。此外,老年小鼠的基因表达模式在各器官之间并不一致。也就是说,心脏中与年龄相关的基因表达模式与大脑中观察到的不同,而大脑中的表达模式与肌肉中的不同,等等。这些结果与衰老不是遗传程序的结果,而是更能反映了组织类型之间差异很大的随机事件的观点相一致。
旨在评估与年龄相关的基因表达模式的研究结果表明,微阵列技术对生物衰老学的有用性可能在于确定延缓衰老的干预措施如何改变基因表达模式。例如,我们现在知道,通过减少热量摄入来保持健康的体重可以显著减缓人类许多器官系统的衰老速度。这些效应可能与年龄相关的基因表达形式的变化直接相关,该研究结果是从热量限制的小鼠的cDNA上得到的。将热量摄入限制在自由获取食物的小鼠正常消耗热量的70%-80%会显著减少所有生理系统的功能损失。(这将在第10章中进一步描述。)自由进食和限制热量摄入的老年小鼠之间的基因表达谱比较显示出巨大差异。尽管基因表达谱的差异因器官而异,这些差异如何直接影响衰老的速度尚待确定,但这些结果提供了基因可以影响衰老和寿命的确凿证据。识别出那些对干预做出反应的特定基因,将会大大提高我们对导致衰老速率遗传差异的因素的认识。